使用seaborn绘制聚类热图
在生物信息领域,无论是基因芯片,RNAseq还是其他各种生信分析里面,差异基因的寻找总是最基础的工作,而热图在差异基因的可视化展示方面占据了独一无二的地位,在这里我将介绍如何使用seaborn库里面的clustermap函数来进行差异基因的可视化展示。
代码
1.导入seaborn等库
import numpy as np
from seaborn import clustermap
import seaborn as sns; sns.set(color_codes=True)
import pandas as pd
2.生成随机矩阵
df1 = pd.DataFrame(np.random.random((1000,10)), columns=["a"+str(i) for i in range(1,11)])#生成随机矩阵1
df2 = pd.DataFrame(np.random.random((1000,10))*2, columns=["b"+str(i) for i in range(1,11)])#生成随机矩阵2
df3 = pd.DataFrame(np.random.random((1000,10))*3, columns=["c"+str(i) for i in range(1,11)])#生成随机矩阵3
#df.rename( index = df["Gene Name"],inplace=True)#设置dataframe的行名
#df.columns=["A1","A2","A3","B1","B2","B3","C1","C3"]#设置dataframe的列名
df = pd.concat([df1, df2], axis=1, sort=False) #合并1,2矩阵
这里生成了三个dataframe,平均值在1,2,3上,每一类使用a,b,c加上数字后缀1到10,使得后面的结果可初步划分为3个大类。
3.绘图
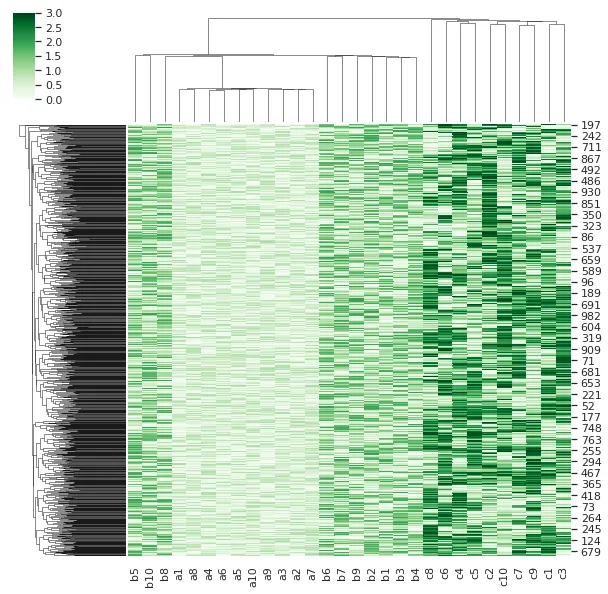
3.1 一种颜色
#设置聚类热图颜色
#1.一种颜色
#如果整个图片只显示一种颜色的梯度的话
g = sns.clustermap(df,cmap="BuPu") #绘图
g = sns.clustermap(df,cmap="Greens") #绿色
3.2 二种对立颜色(红绿)
#设置聚类热图颜色
#如果整个图片显示二种对立颜色的梯度的话
#2.二种对立的颜色
g = sns.clustermap(df,cmap="PiYG")
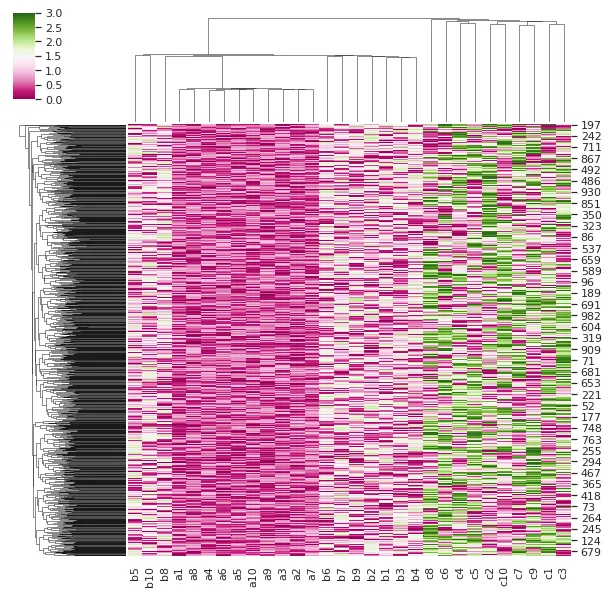
从上面的结果中可以看到热图分为4个区域,其中b区域被a分割到最左边,这是因为这部分的b数值较小,被聚到了a部分,当然大体上是对的(因随机函数问题造成分类不均).
总结:
在clustermap函数里面其实是使用了heatmap的,但是加上了聚类功能,使得其绘制出来的热图具有横向样本(sample)和纵向基因(gene)的聚类功能,更符合生物信息学分析的要求。
在实际应用过程中,在导入差异基因的数据表后,要记得把纵列改为基因的名称(上面注释代码里面有关于pandas里面dataframe中行名和列名的修改)。
以上就是使用seaborn的ClusterMap绘制聚类热图的介绍,如有疑问,多看看官方文档。。
- 原文作者:春江暮客
- 原文链接:https://www.bobobk.com/98.html
- 版权声明:本作品采用知识共享署名-非商业性使用-禁止演绎 4.0 国际许可协议进行许可,非商业转载请注明出处(作者,原文链接),商业转载请联系作者获得授权。